筆者前一陣子有遇到一個分析的案子,這個案子很特別,收集資料的方式是以季為單位,每一個有乾癬診斷的病人,會去收集以乾癬診斷的那一天開始起算,往後追蹤每一季的資料,因此每個人的資料筆數為每三個月一筆,直到2013/12/31或是退保日期(死亡)為止。因此這個資料的筆數相當的驚人,假設某個病人從我們資料中最早的2001/1/1開始往後追蹤,中間都沒發生死亡,因此這個病人最多會有52筆資料(一年有四季*13年的追蹤),實際上,我們這個資料最後的筆數將近400萬筆資料,這應該也稱得上是所謂的大數據(Big data)了吧!
為了幫助讀者理解資料的長相,如下圖所示,每一筆的資料其實是一季的收集,以第一個人為例,這個人在13年的追蹤過程中,共有8筆(8季的資料)。
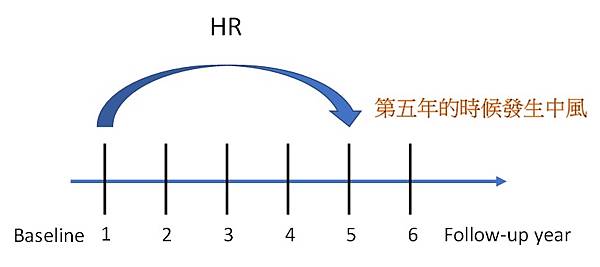
那為什麼我們需要這樣收集資料呢,這個原因其實跟我們的研究目的有關,我們想研究不同生物製劑的暴露對於帶狀泡疹(Herpes zoster)的發生,但傳統世代追蹤研究設計下,收集資料的方式會沒辦法處理暴露會隨著時間有一直改變的情況,通常只收集病人一開始(Baseline)的藥物暴露狀態,藉以觀察與未來outcome(中風)發生的關聯性。如下圖所示,每一個病人所收集的暴露狀態,通常是Baseline一次,然後開始往後觀察,直到發生outcome(中風)。
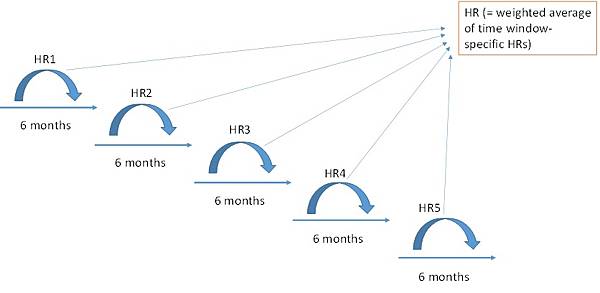
但這時候會遇到一個問題,如果病人使用藥物的暴露狀態會一直改變(第一個月暴露、第二個月不暴露、第三個月暴露),那就有可能發生,其實一個發生outcome的病人在追蹤的過程中其實暴露藥物的機會比起不暴露的時候來得多,但如果只收集Baseline那次的暴露狀態,就有可能因為大多數病人Baseline那次沒有暴露,最後會下結論說此藥物的暴露可能不會增加outcome發生的機會,因此如果要精準的研究藥物暴露與outcome發生的關係,就需要透過下圖的研究設計,透過觀察每一次的暴露區間(Time window),去計算暴露者相較於非暴露者的HR (HR1-HR5),最後把這些HR給合併,計算出一個整體的HR,代表研究期間內藥物真實的暴露情形與outcome之間的關係。
概念講完,開始進入實際操作的部分,首先我們會需要匯入我們的資料到R,筆者這裡提供一個R studio一個匯入資料非常簡單的作法,當我們打開R studio後,我們可以點選Import Dataset,點進去後,如下圖所示,有六種匯入資料格式可以選擇,筆者以SPSS檔案為範例作匯入。
點選完成後,會進入以下畫面,在File/URL的區塊點選Browse,找到您資料放置的位置,這邊要注意一件事情,R不支援路徑當中有出現中文字,如果有中文字會導致匯入資料失敗,路徑確定後,R會直接呈現資料的前50筆在Data Preview這個區塊,同時在右下角Code Preview中,R會自動把匯入資料的語法呈現在上面,讀者可以直接copy這段語法到R studio裡,並執行他,就可以成功匯入SPSS檔到R studio當中,或是讀者也可以直接點選右下角的Import,也可以達到匯入資料的功能,但語法的部分可能就不會留在語法視窗當中。
成功匯入資料後,回到語法視窗,為了要做之後的存活分析,需要先安裝這三個跟存活分析有關的package
library("rms")
library("Hmisc")
library("survival")
把這三個package下載完成並執行後,鍵入以下的語法
data<-data.frame(data) #這邊我會把匯入進來的檔案,通通轉成data frame (R常用的資料格式)
attach(data) #這段主要的功能在於讓R可以直接去讀取資料中的變項,而不需指定是哪一個資料中的變項,例如data$Quarter_start_date
這邊我會特別做參考組的設定,因為我的資料的某些類別變項超過三類,為避免R預設的參考組設定跟我想的不一樣,因此我這邊會特別去指定類別變項的參考組是哪一組
newarea <- factor(newarea) #將資料當中newarea這個變項轉換成類別變項,因為資料原始coding的關係,R會把這個變項當連續變項做分析,因此這邊要手動去調整。
medication_treatment <- factor(medication_treatment)
medication_treatment = relevel(medication_treatment, ref = "14") #ref指的是把medication_treatment=14這組當成參考組
avg_steroid_3gr<- factor(avg_steroid_3gr)
avg_steroid_3gr = relevel(avg_steroid_3gr, ref = "1") #ref指的是把avg_steroid_3gr =1這組當成參考組
完成上述設定後就可以進入最重要的分析環節了,
set.seed (1) #因為存活分析的計算的過程中,可能會有模式迭代計算的問題,因此會設定seed,讓每一次分析的結果維持一樣。
以下的每一段語法都會分為兩個版本,首先我們先執行單變項的time-dependent Cox model,其中語法的組成包含以下項目
Quarter_start_date(每一個人一開始的追蹤日期),TR_new_date(每一個人結束的追蹤日期),Later_HP(Outcome發生有無),~左邊的是依變項的設定,右邊則是自變項的設定,目前只有一個自變項(medication_treatment)。
fit <- coxph(Surv(Quarter_start_date, TR_new_date, Later_HP) ~ medication_treatment, data=data)
fit #印出分析的結果
以下的語法其實跟上面的語法是差不多的,唯一的差別在於最後面加上了”cluster(ID)”,這是甚麼意思呢,其實我們想一下我們分析資料的型態,每一個人其實都會有多筆的資料,也代表一件事是每一個人的每一筆資料其實是相依的資料,同一個人的每一筆資料其實都有關聯性,不是彼此獨立的,因此為了考慮這個關聯性,我們可以加”cluster(ID)”到模式當中。
fit1 <- coxph(Surv(Quarter_start_date, TR_new_date, Later_HP) ~ medication_treatment+cluster(ID), data=data)
fit1 #印出分析的結果
以下的語法是多變項分析的結果,唯一的差別就只差在當我們的自變項不只一個的時候,可以透過”+”,來去加上其他的自變項,就可以得到考慮許多自變項後的分析結果。
fit2 <- coxph(Surv(Quarter_start_date, TR_new_date, Later_HP) ~ male_sex+age_var+newarea+MH_DM+MH_HTN+MH_Lipid+MH_CKD+MH_Gout+MH_PSA+MH_CCI_total_score+
medication_treatment+Statin_m+lN_steroid_cum_dose, data=data)
fit2
fit3 <- coxph(Surv(Quarter_start_date, TR_new_date, Later_HP) ~
male_sex+age_var+newarea+MH_DM+MH_HTN+MH_Lipid+MH_CKD+MH_Gout+MH_PSA+MH_CCI_total_score+
medication_treatment+Statin_m+lN_steroid_cum_dose+cluster(ID), data=data)
fit3
參考資料:
- coxph 介紹: https://www.rdocumentation.org/packages/survival/versions/3.2-3/topics/coxph







 留言列表
留言列表


 {{ article.title }}
{{ article.title }}